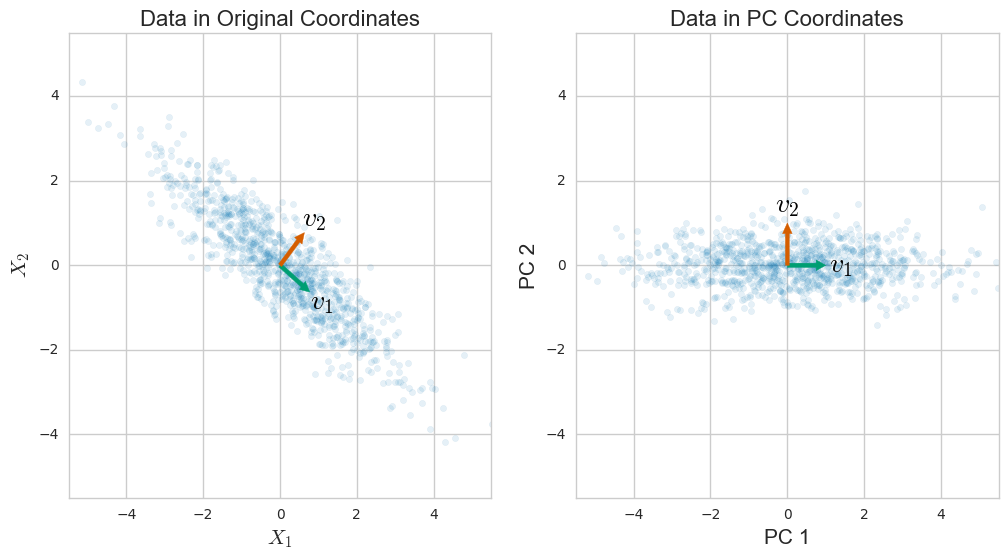
Biarkan saya mulai dengan PCA. Misalkan Anda memiliki n titik data yang masing-masing terdiri dari angka d (atau dimensi). Jika Anda memusatkan data ini (kurangi titik data rata-rata dari setiap vektor data x i ), Anda dapat menumpuk data untuk membuat matriksμxsaya
X= ⎛⎝⎜⎜⎜⎜⎜⎜xT1- μTxT2- μT⋮xTn- μT⎞⎠⎟⎟⎟⎟⎟⎟.
Matriks kovarians
S= 1n - 1∑i = 1n( xsaya- μ ) ( xsaya- μ )T= 1n - 1XTX
S
S= VΛ VT= ∑i = 1rλsayavsayavTsaya,
vsayasayaλsayasayaSsaya
A = ( 1021)kamusayavsaya
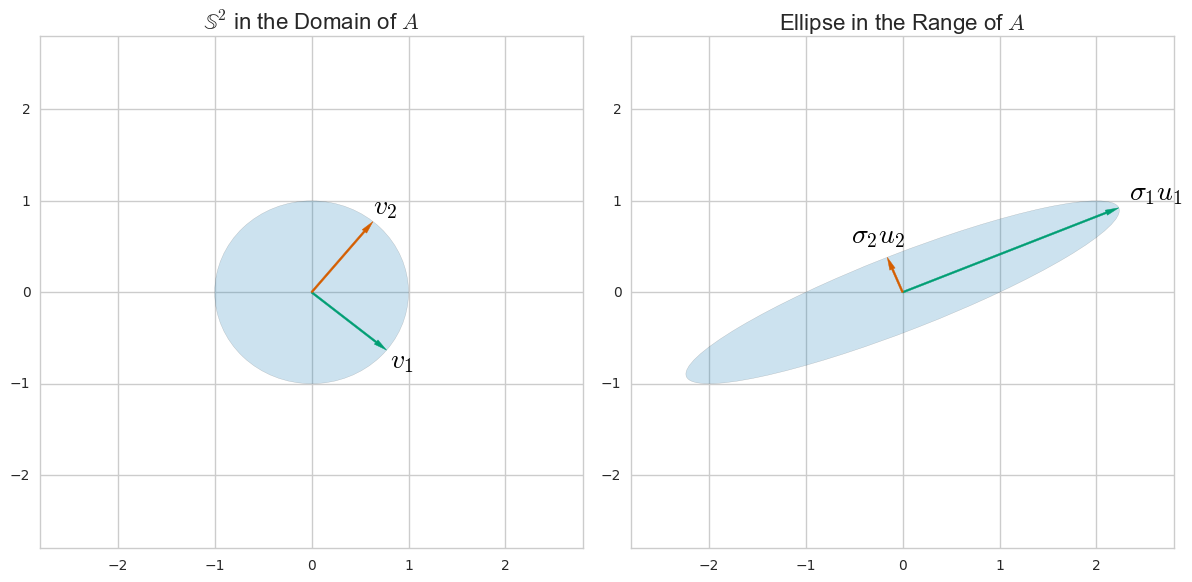
SEBUAHSkamusayavsaya
XA = X
X= ∑i = 1rσsayakamusayavTj,
{ usaya}{ vsaya}Svsaya
kamusaya= 1( n - 1 ) λsaya--------√Xvsaya,
σsaya
σ2saya= ( n - 1 ) λsaya.
kamusayaXkamusayaXsayavsayaX
Saya membahas lebih detail dan manfaat hubungan antara PCA dan SVD dalam artikel yang lebih panjang ini .


